実験データの処理方法について
−入門編−
I. Hidaka
(うぴお会)
Dec. 4, 1997
Updated on Jul 5, 2001
はじめに
この文書は、身体教育学コースの学部生、大学院生を対象に、実験データの処理の基礎をレクチャーする目的で書かれています。スクリプト例文はどなたでも自由に使って頂いて構いませんが、バグがある可能性をお忘れなく。自己責任でご利用ください。
スクリプトの実行環境は、Solaris Workstation (educhan /
complex)上でkshをシェルとし、TSASの各プログラムとgawkが動作可能であることを想定しています。Cygwinを使うと、Windows上でも同様の環境を構築することが可能です。私自身は、Cygwin1.1.7のbashを多用しています。(そのうちLinuxに移行するかもしれませんが。)
目 次
第1章 基本編
第2章 応用編
参考書
第1章 基本編
スクリプトを使ったデータ処理とは?
計測プログラム(collectやreflex、breathなど)で取得したデータから知りたい情報を引き出すためには、生データに対していろいろな数値処理を施す必要があります。例えば、ある区間のデータのみ抽出したり、平均値や標準偏差を計算、帯域フィルタにかける、補間する、FFTにかけて周波数毎のパワーを計算する、などが挙げられます。
これらの処理を行う上でもっとも簡単な処理の方法は、WindowsやMacで動くExcelやOriginなどのGUIを備えたソフトを使い、マウスを使って目で見ながら作業することでしょう。この方法は、処理するデータ量が少なければ、けっこう速いですし、また、そこそこ豊富な関数/ツール群も揃っていますので、非常に便利です。しかし、大量のデータを処理するとなると、同じ操作の繰り返しを延々と続けねばならず、また、途中の処理を変えようと思ったら、全てをやり直さなければなりません。これはきつい。
しかし、シェルスクリプトを使えば、大量のデータファイルを一度に、かつ高速に処理することが可能です。また、一度作ったスクリプトは、別の処理にも転用可能です。
次の例は、Korn
shell (コーンシェル、ksh)を使って複数のデータに対してWigner-Ville分布を求めるスクリプトです。
< test1.ksh > <-----------
以下、<filename>でファイル名を表すこととします。
for b in data/s*in
do
gawk -f rri.awk $b > temp.4
filter /is:0.001 < temp.4 > temp.5
wvdist /sl:4 /is:0.001 /wl:6 /ht /lt /ta:20 /rf < temp.5
> temp.6
gawk -f skip2.awk -v x=5 temp.6 > $b.wvd
rm -f temp.?
done
この文章(スクリプト)を"test.ksh"という名前のファイルに書き込み、これを実行すると、
- dataディレクトリの中にある、名前がsで始まりinで終わるすべてのファイルを、
- gawkというプログラムで行と列を整えて、
- filterというプログラムで、処理しやすいように整形して、
- Wigner-Ville分布を計算し、
- 再度gawkで、今度はgnuplotで三次元描画できるように変形する
という処理が瞬時にできてしまいます(実際にはもう少し下準備が必要ですが...)。シェルスクリプトとは、このようにデータがいろいろなプログラムによって処理されていく流れを記したものです。こうしておくと、手順が複雑な処理でも間違えることはありませんし、処理をやり直すことも簡単です。また、自分が作ったプログラムを組み合わせることもできます。
スクリプトを書くには、個々のコマンドやプログラムの使い方と、プログラムからプログラムへデータを渡す方法、できたデータをファイルに保存する方法などを知っておく必要があります。まずは、基本的なUNIXのコマンドを覚えてください。
UNIXのコマンド
シェルスクリプトでよく使うコマンドを紹介します。
cp a b
ファイルaと同じ物(コピー)をつくり、bという名前をつける。
あるいはファイルaをディレクトリbにコピーする。
あるいはディレクトリaをディレクトリbにコピーする。
mv a b
ファイルaをbという名前に変更する。
あるいはファイルaをディレクトリb内に移動する。
あるいはディレクトリaをbという名前に変更する。
rm -f a
ファイルaを削除する。
mkdir c cという名前のディレクトリを作る。
rm -r c cというディレクトリと、その中のファイル・ディレクトリすべてを削除する。
cd c cという名前のディレクトリに移動する。
cd ..
一つ上のディレクトリに移動する。
echo "$var\n" 変数varの内容を表示する。\nは改行を表す。
# コメント
#以降、改行までの文は実行されないので、コメントとして使えます。
pwd
自分がいまどこのディレクトリにいるかを調べる。(スクリプトではあまり使いませんが...)
他にもいろいろありますが、まずはこれだけ。
kshスクリプトの使い方
1.ファイル名と拡張子
WindowsやMacでは、ファイル名にひらがな、カタカナ、漢字、記号を比較的自由に使えます。しかし、スクリプト処理をするためには、データもスクリプトも、半角アルファベット&半角数字だけの名前を付けてください。使える記号は、「.」「_」「-」程度です。「$」「\」「#」「?」「[」「]」「*」「{」「}」「+」「&」「|」「(」「)」「;」「"」「'」には特別な意味がありますので、ファイル名として使ってはいけません。
データファイルは、スペースかタブで区切られた、各変数が1列をなす形式のテキストファイルがよいです。beatやcollectで測定したデータなら、はじめからこの形式になっています。Excel形式は、他のプログラムでは読めないので使えません。Wordの文書は、確かに「テキスト」ではありますが、これも他のプログラムでは一般に読めないバイナリファイルです。
データファイルの名前は、一定の規則を自分で考えて命名するようにすると、後の処理が非常に楽になります。例えば、s01_ct.1のようにして、被検者番号、プロトコル、試行番号を使う。あるいは、実験条件ごとにディレクトリ(フォルダ)を作製し、そこにデータを格納するという方法もあります。しかし、私は、同時に処理するデータは一つのフォルダにまとめておく方が効率的だと思っています。
Korn Shellのスクリプトファイルは、拡張子を.kshとしてください。実は何でもいいのですが、こうするとすぐにわかりますし、データや他のプログラムと混同することが避けられます。UNIXのコマンドと同名にしないようにしてください。ある名前のコマンドが存在するかどうかは、whichを使うとわかります。
<hidaka@educhan:235>$ which ftspec
/usr/local/bin/ftspec <------------
ftspecはすでに存在。
2.パーミッション
スクリプトファイルを実行するには、まず実行パーミッションを得ておかねばなりません。そのためには、chmodというコマンドを使います。
chmod
+x test.ksh
3.データ読み込み
スクリプトにデータを読み込ませるには、引数(ここではdata1)として与える方法と、スクリプト内で指定する方法の2通りがあります。
方法1: test.ksh datafile1 5 15
方法2: test.ksh
(スクリプトの中でデータを指定)
方法1の場合、datafile, 5, 15 は、スクリプトの中では$1,
$2, $3という名前(変数)に置き換えられます。このスタイルは、1個のデータを処理する場合に使います。
< test2.ksh >
filter /sl:4 /xc:2 /yc:3 /is:0.001 /bg:$2
/ed:$3 < $1 > $1.tilt.dat
方法2では、for文とワイルドカードを使って、条件に当てはまるデータファイルすべてを処理することが多いです。私はこちらをよく使います。
< test3.ksh >
for data
in filtered/*.dat
do
ftspec /sl:4 /is:0.001 /bt:8 /sp:10 /lt /cg < $data |
autons /cg /ai /nz > $data.temp
gawk -f autons.awk $data.temp
>>result.dat
rm -f temp.*
rm -f $data.temp
done
4.リダイレクトとパイプ
スクリプト内では、複数のコマンドによってデータが次々に処理されていきますが、コマンド間でのデータの渡し方には、2通りの方法があります。
(1)データをいったんファイルに保存して、渡す。
このときはリダイレクト "<" , ">" を使います。すでに存在するファイルに対し、リダイレクト
">" でデータを送ると、以前のデータはなくなります。二重のリダイレクト
">>"では、以前のデータの下に、新しいデータが書き足されます。
TSASの各プログラムは、 "<" でデータを入力するものが多いのですが、rowcatのように
"<" を必要としないものもあります。バグとなりやすい部分です。
(2)データをそのまま渡す。
パイプ "|" を使います。
5.ループと変数
(1) for ループ
(1-1) 変数varの部分に、v1、v2、v3...のように順に名前を代入し、それぞれに対してdoからdoneで囲まれた部分(cat
$var)を実行する。varに入れるのは、ファイル名でもただの番号や文字列でも構いません。
< test4.ksh >
for var in v1 v2 v3 v4
do
cat $var
done
この場合には、cat v1というコマンドを実行後、次にcat
v2、cat v3、..の順で実行していきます。.
(1-2) 変数varに、先頭がvで始まるすべてのファイルの名前を代入し、それぞれに対してdoからdoneで囲まれた部分を実行する。varにはファイル名しか使えません。
< test5.ksh >
for var in v*
do
cat $var
done
この場合には、v1, v12, vab, v,
...などが処理の対象となります。「v*」の代わりに「v?」を使うと、v1,
va, v- ...
など、「?」の部分がなんらかの1文字からなるファイルのみが対象となります。「v???」ならば3文字です。
(2) while ループ
< test6.ksh >
integer n
# integer(整数)宣言は、なくても動く。
n=1
# =の前後に空白を入れてはいけない
while (( n < 20 ))
do
cat data.$n
(( n = n + 1 ))
done
whileループでは、while以下の部分が真(すなわち、変数nが20未満)であるかぎり、doからdoneで囲まれた部分を実行しつづけます。注意すべき点は、
1.シェルスクリプトでは整数変数しか使えない(小数点は扱えない)
→実は工夫するとできる。
2.条件判定に使う変数nは、事前に初期化が必要
3.do-doneの中で、nを変化させる操作が必要。
4.数値演算/比較時には(( ))で括ることが必要。
6.条件判定
(1) 数値の比較
test7.kshでは、test.datを10分間の区間毎にfilterにかけます。10,
50,
100分時の区間を処理した時に、メッセージが表示されます。
(注:データ数と分割区間数が非常に多い場合には、毎回filterで全データを読みこむ方法では遅くなるかもしれません。gawkでデータを小分けする手もあります。)
< test7.ksh >
i=0
j=10
while ((i <= 100))
do
filter /bg:$i /ed:$j < test.dat > $i.dat
if (( i == 10 ))
then
echo "10 min\n"
elif (( i == 50 ))
then
echo "50 min\n"
else (( i == 100 ))
echo "100 min\n"
fi
# elseにthenは不要
(( i = i + 10 ))
(( j = i + 10 ))
done
(2) 文字列の比較
被検者aのみ、分析区間を変更します。
< test8.ksh >
for subject in a b c d e
do
if [[ subject = a
]] #
[[ と ]]の間の要素間は、必ず空白で区切ること。
then #
文字列の場合、=で比較するが、==でも動く?
bg=3
ed=13
else
bg=1
ed=11
fi
filter /bg:$bg /ed:$ed < $subject > fil/$subject
done
Korn Shellの全機能を解説するときりがないので、あとは参考書を見てください。
TSASの使い方
TSASとは、時系列データ解析用のプログラム群の総称です。
TSASのプログラムのうち、よく使われる
"filter", "ftspec", "rowcat"の機能と使用法を以下に紹介します(TSAS日本語マニュアルより抜粋、改変)。その他のプログラムについては、
マニュアル: ホームページ版や印刷版
オンラインヘルプ: man tsas と入力
各プログラムのヘルプ: ftspec /he と入力
を参照してください。
1. filter
filterは、生データ中のデータの選択、異常値の除去、補間といった前処理を行うプログラムです。まずは、心電図R-R間隔時系列データをfilterで処理してみます。
○ 入力ファイル(beat1):beat.exeで測定。EXT2はティルト角度。
Time RR SBP DBP EXT1 EXT2
NCP1 NCP2 TOTAL
0.02 1385 135.4 67.0 10.68 -0.26 -0.9 4.91 1
0.05 1510 133.0 63.1 6.28 -0.26 0.0 4.91 2
0.07 1424 131.1 62.6 7.61 -0.26 0.0 5.02 3
0.09 1154 127.7 66.5 8.95 -0.26 0.0 4.91 4
0.11 1188 127.2 65.0 4.01 -0.26 0.0 4.91 5
....................................
16.86 702 89.8 55.3 3.07 60.52 0.0 5.02 1186
16.87 679 88.8 55.8 4.14 60.26 0.0 5.12 1187
16.88 695 89.8 55.8 6.68 60.52 0.0 5.02 1188
○ 使用スクリプト:
filter /sl:1 /xc:2 /yc:6 /is:0.001 /ip:3,4.0 /bg:0.0 /ed:15.0 /fa
< beat1 > beat1.fil
ここでは、入力データの2行目以降、第2列(RRI,
ms)と第6列について、0分から15分までのものをマニュアルで異常値除去を行い、3次スプライン関数で補間して4Hzで再サンプリングするように支持しています。
○ 処理中の画面
カーソルキー等を使って、異常値を除去できます。
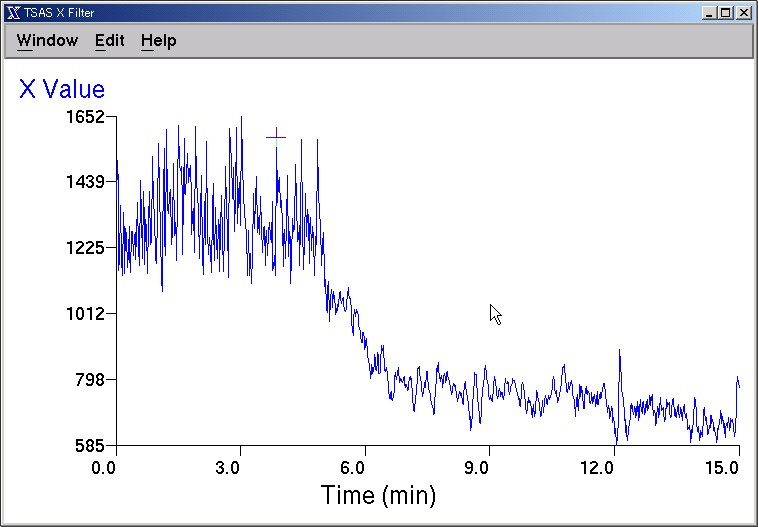
○ 出力ファイル(beat1.fil):
Ndata = 3594
Xmean = 951.61139 Ymean = 40.06742
Xs.d. = 278.18707 Ys.d. = 28.59848
Time X
Y
0.02308 1389.81042 -0.26000
0.02725 1405.84766 -0.26000
0.03142 1418.93774 -0.26000
0.03558 1429.23962 -0.26000
0.03975 1436.91248 -0.26000
..................
14.98540 793.00000 60.52000
14.98956 780.47424 60.51991
14.99373 771.33331 60.52000
TSASの各プログラムは、上記のように、/x:y
というスタイルのオプションを列記することによって、行いたい処理を指定します。filterの詳細な使い方は、以下の通り。
|
Usage: filter [option]
<infile >outfile
Option: /SL:n
/XC:n and/or /YC:n
/IS:fi or /TS:ft or /TF
/IP:n,f
/BP:n,fl,fu
/BG:f
/ED:f
/FA or /FX:f,f and/or /FY:f,f
/FB
/MQ
/LT
/SF
/SV
/HE
filter はデータ列と時間のデータを選択し、必要があれば異常値をグラフィカルな画面上で修正・補間・再サンプリングし、以降のデータ解析に備えます。
入力ファイルとの形式は、
<infile>
header 1
header 2
....
header nh
###.### ###.### ###.### ....
###.### ###.### ###.### ....
.....
というものを想定しています。ヘッダが何行かあり、データは列でまとめられています。
filterの出力ファイルは
<outfile>
Ndata = ##########
Xmean = ####.##### (Ymean = ####.#####)
Xs.d. = ####.##### (Ys.d. = ####.#####)
Time X (Y)
####.##### ####.##### (####.#####)
####.##### ####.##### (####.#####)
.....
ここで、Ndataはデータ数、XmeanはXの平均値、Xs.d.はXの標準偏差を表します。以下同じ。
オプションの機能とデフォルトの設定は、、
- /SL:n
- 読み込まれたデータの最初のn行をスキップします。上の例では、/SL:nhによりヘッダをスキップし、データを先頭から読み込みます。デフォルトではn=0。
- /XC:n and/or /YC:n
- 二つの時系列XとYが第何列にあるかを指定します。時系列Yは存在しなくてもかまいません。デフォルトでは/XC:1、/YC:2。
- /IS:fi or /TS:ft or /TF
- データXがデータの間隔を表している場合(/IS)、一定の時間間隔でサンプルされている場合(/TS)、breathの出力ファイルのように等間隔でない時刻を表している場合(/TF)とで使い分けます。
-
- /ISオプションでは、時間間隔Xの単位を秒に換算するための乗数fiを指定します。つまり、時間間隔がms単位で測定されている場合には、fiは0.001となります。
-
- /TSオプションでは、サンプリング周波数ft(Hz)を指定します。
-
- デフォルトでは、/TS:1.0。単に/ISとのみ指定した場合には、fi
= 1.0。
- /IP:n,f
- 等間隔・不等間隔の入力データに対し、補間を施します。nは補間の方法で、n=0で0次ホールド、n=1で直線補間、n=3で3次混合スプライン補間を行います。この値以外のnが指定されていた場合、自動的にn=0がセットされます。fは新たなサンプリング周波数で、出力データはこの周波数でサンプリングされた等間隔のデータとなります。
- /BP:n,fl,fu
- FIR型デジタル帯域フィルタをかけます。nはフィルタの次数で、デフォルトはm=20。flおよびfuは、それぞれ低周波及び高周波側の遮断周波数で、両者ともナイキスト周波数に対する割合で与えます。デフォルトはfl=0.0、fu=1.0の全域通過フィルタです。
- /BG:f
- データの分析を始める時刻(分)を指定します。デフォルトではf=0.0。
- /ED:f
- データの分析を終える時刻(分)を指定します。デフォルトでは入力ファイルの最後に相当する時刻となります。
- /FA or /FX:f,f and/or /FY:f,f
- /FAを指定すると、時系列解析の結果に影響を与える異常値をマニュアルで除去します。指定しなければ、除去しません。以前のバージョンでは、/FX,
/FYオプションを指定することによって自動補間が可能でしたが、最新版ではこの機能はありません。
-
- マニュアル除去時において、/TSオプションが指定されていると、隣接するデータからの一次補間が行われます。/ISオプション時には、タイムスケールを変えないような新しいインターバルが挿入されます。
- /FB
- 第1列がタイムスタンプとなっているFirst
Breath社の呼吸・ガス交換測定プログラムに対して用いられます。この形式でないデータに対してこのオプションを指定すると、以後のプロセスはうまくいきません。
- /MQ
- Marquette社のHolter心電図記録計のデータに対して用いられます。/MQを指定する前に、データをmar2filにより前処理する必要があります。
- /LT
- データからリニアトレンドを除去します。デフォルトでは指定されません。
- /SF
- 出力を浮動小数点による科学形式で行います。変動が小さいデータを処理する際には、このオプションの使用をおすすめします。デフォルトでは指定されません。
- /SV
- 実行時にバージョン情報を画面に出力しないようにします。
- /HE
- 使用可能なオプションの一覧を画面に表示します。
|
2. ftspec
FFTによりスペクトルを計算するプログラムです。スペクトルにおける様々なオプションを指定できます。先程のデータのうち、0-5分の区間のRRI,
SBPの自己スペクトルとクロススペクトルを計算してみます。
○ 使用スクリプト
filter /sl:1 /xc:2 /yc:3 /is:0.001 /ip:3,4.0 /bg:0.0 /ed:5.0 < beat1 >
beat2.fil
ftspec /sl:4 /ts:4.0 /bt:9 /sp:5 /lt /wb /fa:3 < beat2.fil > beat2.spc
○ 出力ファイル(beat2.spc):
Ndata = 1191
Xmean = 1317.94421 Ymean = 111.19390
Xs.d. = 93.63486 Ys.d. = 12.61414
Freq Sxx Syy
|Sxy| Phase Coh
0.00000 341.49680 38.83525 7.99765 0.00000 0.00482
0.00781 410.63696 14.98144 7.22176 -2.40392 0.00848
0.01562 331.77554 3.58977 6.07027 -1.38572 0.03094
0.02344 304.17261 4.86989 10.00387 -1.76313 0.06756
..........................................
1.97656 0.01147 0.00000 0.00011 -1.38156 0.37431
1.98438 0.00955 0.00000 0.00008 -0.43519 0.30410
1.99219 0.00955 0.00000 0.00008 -0.43519 0.30410
SxxはRRIの、SyyはSBPの自己スペクトルです。SxyはSBP->RRIのクロススペクトルです。
○ スペクトル
0.25Hzでのピークは呼吸性不整脈によるものです。
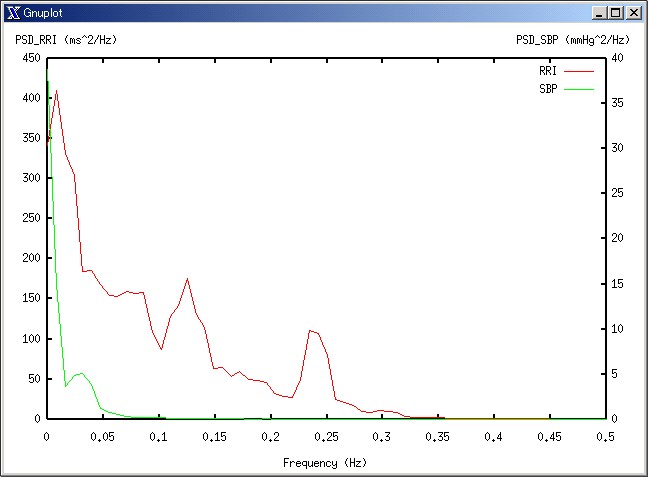
○ 使用法:
|
Usage: ftspec [option]
<infile >outfile
Option: /SL:n
/IS:fi or /TS:ft
/BT:n
/SP:n
/LT
/CG:n
/WB or /WH
/FA:n
/RF or /RT or /RI:n
/SF
/SV
/HE
ftspecは、Cooley-TukeyのFFTアルゴリズムを用いて、自己スペクトル密度とクロススペクトル密度(それぞれASD、CSD)、自己相関関数と相互相関関数(ACF、CCF)を計算します。
入力ファイルの形式は、filterの出力ファイルと類似のフォーマットを用います。
<infile>
Ndata = ##########
Xmean = ####.##### (Ymean = ####.#####)
Xs.d. = ####.##### (Ys.d. = ####.#####)
Time X (Y)
####.##### ####.##### (####.#####)
####.##### ####.##### (####.#####)
.....
出力ファイルの形式は、使用したオプションにより異なります。
</RF オプション使用時のoutfile>
Ndata = ##########
Xmean = ####.##### Ymean = ####.#####
Xs.d. = ####.##### Ys.d. = ####.#####
Freq Sxx Syy |Sxy| Phase (*)Coh
###.#### ###.#### ###.#### ###.#### ###.#### ###.####
###.#### ###.#### ###.#### ###.#### ###.#### ###.####
......
Freqは周波数(Hz)、Sxxは変数Xの自己スペクトル密度、Syyは変数Yの自己スペクトル密度、|Sxy|は変数X、Y間のクロススペクトル密度のベクトルの長さ、Phaseは変数X、Yの位相差、CohはX、Y間の2乗コヒーレンスを表します。ただし/CGオプションを使用した場合には、|Sxy|の代わりに変数Xの粗視化スペクトル(CGSxx)が書き込まれます。
</RT オプション使用時のoutfile>
Ndata = ##########
Xmean = ####.##### Ymean = ####.#####
Xs.d. = ####.##### Ys.d. = ####.#####
Lag Cxx Cyy Cxy+ Cxy-
###.#### ###.#### ###.#### ###.#### ###.####
###.#### ###.#### ###.#### ###.#### ###.####
......
Lagは相関関数におけるラグ(秒)、Cxxは変数Xの自己相関関数、Cyyは変数Yの自己相関関数、Cxyは変数XからYへの相互相関関数をあらわします。/CGオプションが指定された場合には、/RTオプションは機能しません。
</RI オプション使用時のoutfile>
Ndata = ##########
Xmean = ####.##### Ymean = ####.#####
Xs.d. = ####.##### Ys.d. = ####.#####
Lag Cxx Cyy Ixy+ Ixy-
###.#### ###.#### ###.#### ###.#### ###.####
###.#### ###.#### ###.#### ###.#### ###.####
......
/RI:1ならIxy+, Ixy-が、/RI:2ならIyx+, Iyx-が出力されます。+はラグt>0の応答、-はt<0の応答です。
オプションの機能とデフォルト値は、
- /SL:n
-
- /IS:fi or /TS:ft
-
- /BT:n
- FFTのビット数を指定します。デフォルトでは、FFTを行うデータ長として、読み込んだデータ長の90%以内でもっとも大きい2のべき乗のデータ点がFFTの対象となります。
- /SP:n
- データを分割して作るサブセットの数を指定します。この操作は、推定されるスペクトルの変動係数(CV)を減少させるために行われます(/FAオプションも参照して下さい)。デフォルトでは、/CGオプションが指定されていなければ、n=3、/CGオプションが指定されていればn=10。
- /LT
- 低周波域のスペクトルに大きな影響を与えるリニアトレンドを、入力データから除去します。この操作はFILTER.EXEでも行えます。デフォルトでは指定されません。
- /CG:n
- 通常のスペクトル解析に加え、粗視化スペクトル解析(CGSA)[1,2]を行います。注)/CGオプションを指定すると、Syy、Gain、Phase、Cohに代わって、変数Xの粗視化スペクトルと、それに関連した変数が出力されます。nはCGSAを行う列の番号を指定します。nを2(デフォルト)以外に指定すると、その列番号に相当するデータ列のCGSAが行われます。もし変数YのCGSAを行いたいのであれば、n=3と指定します。(/ISオプションが指定されていれば、時間のデータは第2列のデータ列から読みとられます。)FTSPEC.EXEは、FILTER.EXEの出力ファイルを入力ファイルに想定しています。したがって、nを2、3以外に指定すると正しく動作しません。デフォルトでは指定されていません。
- /WB or /WH
- これらのオプションでは、ウィンドウの種類を指定できます。/WBオプションではBinghamのコサイン型ウィンドウが、/WHではHanningのウィンドウが選択されます。デフォルトは/WBです。
- /FA:n
- このオプションは/SPオプションとともに、推定されたスペクトルの変動係数を少なくするために用いられます。nは周波数域でのスペクトル平均化の次数(スペクトル・ウィンドウ)を表しています。/SPオプションと/FAオプションを併用すると、推定されたスペクトルの変動係数CV(%)は、
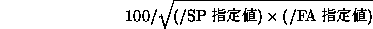
に減少します。デフォルト値はn=1です。
- /RF or /RT or /RI:n
- このオプションは、出力形式を指定する際に用いられます。/RFオプションは周波数領域から分析した結果を、/RTオプションは時間領域から分析した結果を出力し、/RIオプションでは周波数伝達関数からインパルス応答を求めます。周波数伝達関数は、クロススペクトルを入力の自己スペクトルで除したものです。したがって、n=1と指定してXで割れば、Xにインパルスが入った場合のYの応答を求めることになります。n=2ならばYへインパルスを入力したときのXの応答です。デフォルトは/RF。
- /SF
-
- /SV
-
- /HE
|
3. rowcat
異なるファイルの列と列を並べて出力するプログラムです。よく使います。
|
Usage: rowcat [option] files
>outfile
Option: /SL:n
/XC:n
/SF
/SV
/HE
時として、TSASの複数の出力ファイルから、ある特定の列だけ抜き出し、それらをひとつのファイルにまとめる必要がでてきます。このような場合に、ROWCAT.EXEを使用して下さい。例えば、個々の出力ファイルを求める場合、異なるファイルの特定の列をくっつける場合、SASなどを使って出力ファイルを検定する場合などにROWCAT.EXEが役に立ちます。
<infile>
header 1
header 2
....
header nh
###.### ###.### ###.### ....
###.### ###.### ###.### ....
.....
出力ファイルのフォーマットは、以下の通りです。
<outfile>
X(1) X(2)
####.##### ####.##### ....
####.##### ####.##### ....
......
各オプションの機能とデフォルト値は、以下の通りです。
- /SL
-
- /XC:n
- このオプションは、入力ファイルから抜き出す列の番号を指定します。デフォルトは、/XC:1です。
- /SF
-
- /SV
-
- /HE
|
gawkの使い方
1.どんなときに使うか?
TSASは非常に強力なツールですが、TSASだけではできないこともあります。例えば...
(例1:)
Labpackの出力ファイルなら、そのままfilterに入れることができます。しかし他の測定プログラムのデータは、そのままではTSASで処理することができない場合があります。例えば、磁気センサによる位置測定プログラムFASTRAKの出力は、
16 03 20.44 13.93 59.60 71.58 72.08 -3.83
32 04 39.28 11.72 61.42 103.09 70.52 -1.31
47 01 34.25 16.99 -45.25 118.09 64.69 28.38
63 02 30.73 16.59 3.04 -25.44 77.82-116.01
79 03 20.43 13.92 59.61 71.69 72.11 -3.74
94 04 39.26 11.74 61.42 103.14 70.53 -1.29
110 01 34.26 16.96 -45.24 118.10 64.79 28.26
125 02 30.72 16.58 3.05 -25.56 77.83-116.19
これは、約16msec周期で4つのセンサの座標とレファレンス点からの角度を順番に記録しています。このデータから、センサ1のX座標とセンサ2のx座標との関連を調べるためには、をTSASで処理するためには、各センサ毎の座標ファイルを作り、適当なサンプリング周波数で補間して時刻をそろえたデータを作る必要があります。
Time Sen_1 Sen_2 Sen_3 Sen_4
0.00104 34.25095 30.72624 20.42781 39.26796
0.00130 34.25192 30.72433 20.42592 39.26524
0.00156 34.25271 30.72260 20.42244 39.26283
0.00182 34.25334 30.72102 20.41785 39.25889
0.00209 34.25425 30.71958 20.41380 39.25471
0.00235 34.25517 30.71711 20.40861 39.25200
0.00261 34.25627 30.71419 20.40419 39.24931
0.00287 34.26114 30.71193 20.39829 39.24715
(例2:)
ある周波数でティルトテーブルをoscillateしたときの心拍変動のスペクトル
Ndata = 1438
Xmean = 1025.96643
Xs.d. = 92.28445
Freq Sxx S2.0 |Sxx'| S0.5 CGSxx
0.00000 47.73786 0.01504 47.73786 0.00786 0.00000
0.00391 211.00533 295.53473 211.00533 290.93399 0.00000
0.00781 356.02304 84.91195 106.86591 134.49606 249.15714
0.01172 80.87994 94.23409 80.87994 95.96165 0.00000
0.01563 53.96445 49.17957 53.96445 141.52199 0.00000
0.01953 65.88317 37.31451 40.58358 44.13905 25.29959
0.02344 196.56624 111.85400 84.76230 64.23237 111.80394
0.02734 92.81770 45.47598 40.38119 35.85719 52.43650
これから、ある周波数+/-0.01Hzの帯域のS/N比を求めるにはどうする??
以上のような、データの整形と計算が必要な場合によく使われるツールが、awk
(gawk) です。
2.gawkの起動
gawkは、
- コマンドラインからてくてく打って使う。
- gawk専用のスクリプトファイル(Korn Shellのスクリプトとは違います)を作っておく。
- kshスクリプト内にgawkのスクリプトを記述しておく。
の3通りの使い方が可能です。(1)だと、どのような処理をしたかの記録が残りませんし、他のコマンドと連携することが出来ません。(2)または(3)をマスターしてください。おすすめは、(3)です。その訳は、ここを参照。
3.gawkスクリプトの実際
awkはプログラミング言語です。すべてを紹介することはとうていできませんので、例をいくつか...
(例1:)さきほどの例1の赤字部分の回答です。gawk -f step1.awk -v number=04 sample.dat
と入力します。step1.awkの中身は以下のようになっています。変数numberを外部から与えています。gawkでは通常スペースやタブを区切り文字として使っていますが、このデータの場合スペースで桁をそろえてあり、所によって異なるデータがくっついていますので、スペース・タブを区切りとして使わず、第何桁から第何桁を読みとれ、という命令を使いました。
< step1.awk >
BEGIN{
FS = "a"
}
{
if(substr($0, 13, 2) == number){
time = substr($0, 1, 10) / 60000
x = substr($0, 15, 8)
y = substr($0, 23, 7)
z = substr($0, 30, 7)
printf("%e %e %e %e\n", time, x, y, z)
}
}
○別解 - (3)の方法 -
< oooo.ksh >
number="04"
:
gawk '{
(この部分は同じ)
}' number=$number sample.dat > sample.ms
:
(例2:)例2の回答です。変数freqは外部から与えています。#はコメントです。
# sn_ratio.awk
# ftspecの出力からOT周波数でのS/N比を算出。
BEGIN{
signal = 0
noise = 0
}
{
if (($1 >= freq - 0.01) && ($1 <= freq + 0.01)){
if ($6 > signal){
name = FILENAME
signal += $6
noise += $4
}
}
}
END{
if (noise > 0){
printf("%s %f %f\n", name, signal, sqrt(signal / noise))
} else {
printf("Noise = 0\n")
}
}
○別解 - (3)の方法 -
< oooo.ksh >
freq="0.03"
:
gawk '{
(この部分は同じ)
}' freq=$freq sample2.dat > sample2.ms
:
このように、gawkは、データを1行毎に読み込んで何らかの処理をし(あるいはせず)、それが終わったら次の行に移ってまた処理をする、というかたちでデータを整形・集計します。正規表現や配列を使うとより多様な処理を行うことができます。
4.Korn Shellスクリプトとの連携
上の(例1:)の処理を、data1, data2, data3,
data4というファイルに対し、センサ01, 02, 03, 04ごとに行うスクリプトは、(2)の方法を使うと、
for sensor in 01 02 03 04
do
for data in data1 data2 data3 data4
do
gawk -f test.awk -v number=$sensor $data > $data.$sensor
done
done
となります。(1)のようにコマンドラインからこの処理を実行するには、16回似たような作業を行わなければなりませんが、このスクリプトであれば1回実行するだけです。
また、こんなつかいかたもできます。
filter /sl:4 /xc:3 /yc:3 /ts:$(gawk -f ts.awk sample1.dat) < sample1.dat > temp.8
ここではsample1.datを一旦ts.awkというスクリプトで処理し、出てきた数値をfilterのオプション指定値に使用しています。事前に計算して紙に書き留めて、手入力でfilterを起動するやり方では、データファイルが多いと大変ですが、gawkとTSAS、kshを組み合わせれば、瞬時に終わります。
その他スクリプト型のプログラムとの連携
1.「連携」とは?
gawkは、kshスクリプトの内部に、
gawk '{
..................
}' var=$var infile
という形式で処理スクリプトを直に書き込み、他のコマンドやプログラムと連携して使うことが可能でした(3
の方法)。しかし、gnuplotやSAS、mathematicaでは、処理を記述した別のスクリプト(gawkでいえば、oooo.awk
というファイルのこと)があらかじめ存在する事を前提としている(2
の方法)ので、このようなスタイルは使えません。
もちろん、それらのソフト用スクリプトを事前に作っておいても構わないわけですが、処理量が多くなってくると、どのスクリプトをどのkshで使っていたか、混乱してきます。そこでこの章では、それらの処理スクリプトをkshスクリプト内で動的に生成/消去する方法を解説します。
要は、echoコマンドで別スクリプトファイルを作ってやるだけです。gnuplotの場合を例に挙げます。
<< 例 >>
#! /bin/bash
fname="test2.ps"
gpstring="
set terminal postscript portrait enhanced color solid 10
set output \"$fname\"
plot sin(x)"
echo "$gpstring" > test.plt
gnuplot test.plt
(注) echo / printf
で、一行一行スクリプトファイルに出力することもできます。
(to be continued)
第2章 応用編
呼吸生理
サンプル:"sample1"
TIME INF VI VE VI+VE VT BF VO2 VCO2 PETO2 PETCO2 RER HR EET13C
0.07 0 16 16 32 1.98 16.62 0.3 0.2 118 29.494 0.89 105.3 9.33
0.13 0 16.3 16.3 32.6 1.39 16.48 0.24 0.2 118 29.46 0.98 109.1 9.42
0.25 0 16.2 16.2 32.4 2.82 8.26 0.22 0.21 122.5 27.554 1.78 101.7 9.44
0.38 0 16.3 16.3 32.6 2.11 7.75 0.163 0.15 125.3 28.108 2.32 105.3 9.37
0.44 0 15.8 15.8 31.6 0.85 16.3 0.277 0.22 124.6 28.905 1.13 107.1 9.31
0.5 0 15.3 15.3 30.6 1.55 15.71 0.325 0.25 124.6 28.94 1.19 110 9.33
0.57 0 18.2 18.2 36.4 1.24 16.09 0.223 0.22 126.3 27.588 1.43 110.2 9.39
0.62 0 17.6 17.6 35.2 1.02 17.34 0.246 0.24 126.7 27.207 1.19 103.4 9.39
0.69 0 16.8 16.8 33.6 1.03 16.26 0.241 0.24 127 27.172 1.36 98.4 9.41
0.75 0 17.9 17.9 35.8 1.05 17.05 0.221 0.22 127.7 26.722 1.3 105.3 9.28
このようなデータから、
- EET13C を入力、 VEを出力とみたときのインパルス応答(60-75分)
- PETCO2 を入力、VEを出力とみたときのインパルス応答(60-75分)
を計算します。手順は、
- 元データからEET13CとVEのセットを、filterを用いて抽出します。その際、3次スプライン関数で補間して、4Hzで再サンプリングします。
- 同様に、PETCO2とVEのセットを作ります。
- ftspecで、EET13Cにインパルスが加えられた時のVEの応答を調べます。
- 同様に、PETCO2にインパルスが加えられた時のVEの応答を調べます。
スクリプトは、このようになります。
filter /sl:1 /xc:14 /yc:4 /tf /ip:3,4 /bg:60 /ed:75 < sample1 > temp.1
filter /sl:1 /xc:11 /yc:4 /tf /ip:3,4 /bg:60 /ed:75 < sample1 > temp.2
ftspec /sl:4 /ts:4 /lt /ri:1 < temp.1 > eet13c_ve.ir
ftspec /sl:4 /ts:4 /lt /ri:1 < temp.2 > petco2_ve.ir
rm -f temp.?
磁気センサ
サンプル:"sample2"
16 03 20.44 13.93 59.60 71.58 72.08 -3.83
32 04 39.28 11.72 61.42 103.09 70.52 -1.31
47 01 34.25 16.99 -45.25 118.09 64.69 28.38
63 02 30.73 16.59 3.04 -25.44 77.82-116.01
79 03 20.43 13.92 59.61 71.69 72.11 -3.74
94 04 39.26 11.74 61.42 103.14 70.53 -1.29
110 01 34.26 16.96 -45.24 118.10 64.79 28.26
125 02 30.72 16.58 3.05 -25.56 77.83-116.19
141 03 20.41 13.91 59.62 71.70 72.13 -3.80
157 04 39.25 11.78 61.41 103.24 70.55 -1.25
172 01 34.25 16.90 -45.27 118.20 64.89 28.27
188 02 30.71 16.58 3.05 -25.61 77.82-116.32
このデータから、センサ1(首)とセンサ2(腰)の前後方向の変化の様子をスペクトル解析します。手順は、
- 各センサごとに、X座標のデータを抜き出します。
- センサが信号を拾う平均周波数を求め、その周波数で補間&再サンプリングします。
- 再サンプリングしたデータの、開始時刻が一致するように先頭部分を適宜切り取ります。
- x座標のデータをまとめます。
- まとめたものからセンサ1とセンサ2のデータのセットを作ります。
- ftspecにより、自己スペクトル(トータル、ハーモニック、フラクタル)とクロススペクトルをもとめます。
<Korn Shell スクリプト>
for sensor in 01 02 03 04
do
gawk -f s_1.awk -v number=$sensor sample2 > tmp1.$sensor
done
s_freq=$(gawk -f s_2.awk sample2)
filter /sl:0 /xc:2 /tf /ip:3,$s_freq < tmp1.01 | filter /sl:5 /xc:2 /tf > tmp2
filter /sl:0 /xc:2 /tf /ip:3,$s_freq < tmp1.02 | filter /sl:4 /xc:2 /tf > tmp3
filter /sl:0 /xc:2 /tf /ip:3,$s_freq < tmp1.03 | filter /sl:7 /xc:2 /tf > tmp4
filter /sl:0 /xc:2 /tf /ip:3,$s_freq < tmp1.04 | filter /sl:6 /xc:2 /tf > tmp5
rowcat /sl:4 /xc:1 tmp2 > tmp6
rowcat /sl:4 /xc:2 tmp2 > tmp7
rowcat /sl:4 /xc:2 tmp3 > tmp8
rowcat /sl:4 /xc:2 tmp4 > tmp9
rowcat /sl:4 /xc:2 tmp5 > tmp0
rowcat /sl:1 tmp6 tmp7 tmp8 tmp9 tmp0 > tmp_a
filter /sl:1 /xc:2 /yc:3 /tf < tmp_a > tmp_b
ftspec /sl:4 /ts:$s_freq /bt:10 /sp:10 /lt /fa:5 < tmp_b > sample2.csd
ftspec /sl:4 /ts:$s_freq /bt:10 /sp:10 /lt /cg:2 /fa:5 < tmp_b > sample2.cgx
ftspec /sl:4 /ts:$s_freq /bt:10 /sp:10 /lt /cg:3 /fa:5 < tmp_b > sample2.cgy
rm -f tmp*
<awkスクリプト>
# s_1.awk
#
BEGIN{
FS = "a"
}
{
if(substr($0, 13, 2) == number){
time = substr($0, 1, 10) / 60000
x = substr($0, 15, 8)
y = substr($0, 23, 7)
z = substr($0, 30, 7)
printf("%e %e %e %e\n", time, x, y, z)
}
}
# s_2.awk
#
BEGIN{
start = 0
end = 0
number = 0
}
{
if(NR == 1){
start = substr($0, 1, 10)
number = 1
} else {
end = substr($0, 1, 10)
number++
}
}
END{
printf("%f\n", 1000 * number / (end - start))
}
COP
サンプル:"sample3"
足圧中心の軌跡を、前後・左右方向ごとにサンプリング周波数100Hzで3回測定しました。
1.010100 -2.022700
1.004100 -2.065000
1.011400 -2.093600
1.023400 -2.122600
1.062600 -2.132800
1.055300 -2.157300
1.067900 -2.164900
1.095300 -2.161400
1.081300 -2.158700
1.087900 -2.140400
1.093900 -2.128400
1.076600 -2.105100
このデータに対し、次のような処理を考えます(以下、KH君のメールの引用です。)。
- 生データにフィルターをかける
- フィルターをかけたデータにFFTをかける
- 求められたスペクトルを区間積分してそれぞれの周波数帯のパワーを求める。
#!/bin/ksh
#usage: psd.ksh *.cop *.cop *.cop
rm tmp*
rm tp*
name=${1%??.cop}
print $name
#spectral analysis of cop
filter /ts:100 /sf /xc:1 /yc:2 <$1 >tmp1
filter /ts:100 /sf /xc:1 /yc:2 <$2 >tmp2
filter /ts:100 /sf /xc:1 /yc:2 <$3 >tmp3
#normal fft
ftspec /sl:4 /ts:100 /bt:10 /sp:6 /sf <tmp1 >tmp4
ftspec /sl:4 /ts:100 /bt:10 /sp:6 /sf <tmp2 >tmp5
ftspec /sl:4 /ts:100 /bt:10 /sp:6 /sf <tmp3 >tmp6
#cgsa X
ftspec /sl:4 /ts:100 /bt:11 /cg:2 /sf <tmp1 >tmp10
ftspec /sl:4 /ts:100 /bt:11 /cg:2 /sf <tmp2 >tmp11
ftspec /sl:4 /ts:100 /bt:11 /cg:2 /sf <tmp3 >tmp12
#cgsa Y
ftspec /sl:4 /ts:100 /cg:3 /bt:11 /sf <tmp1 >tmp13
ftspec /sl:4 /ts:100 /cg:3 /bt:11 /sf <tmp2 >tmp14
ftspec /sl:4 /ts:100 /cg:3 /bt:11 /sf <tmp3 >tmp15
#for ensemble average
ensavg /sl:4 /xc:1 /yc:2 /sf tmp[4-6] > tmp7
ensavg /sl:4 /xc:1 /yc:3 /sf tmp[4-6] >> tmp7
ensavg /sl:4 /xc:1 /yc:4 /sf tmp10 tmp11 tmp12 > tmp16
ensavg /sl:4 /xc:1 /yc:4 /sf tmp13 tmp14 tmp15 >> tmp16
gawk -f psd21.awk tmp7 > $name.fft
gawk -f psd21.awk tmp16 > $name.cgsa
rm tmp*
変数$1から最後の??.copという部分を取り除いた文字列を表示して、処理に取りかかっています。
"tmp[4-6]" は、"tmp4 tmp5 tmp6"という文字列を表しています。
ftspecの出力ファイル3つを、ensavgによりアンサンブル平均を取っています。SxxとSyy、|Sxx'|と
|Syy'を続けて( >>
)書き込んでいるために、次の処理が難しくなっています。rowcatをつかって、
Freq Sxx Syy Freq |Sxx'| |Syy'|
: : : : : :
の形にすると、gawkでの処理がしやすくなると思います。|
心拍変動
サンプル:"sample4"
最後に、長時間の心電図R-R間隔のデータに対し、短時間フーリエ変換(STFT)によって自律神経系の指標が時間とともにどのように変わるかを見てみます。
1086
1050
1106
1118
1055
1091
1104
1058
1122
1156
1080
- RRIがならんだだけのデータをfilterで読み込み、補間して5Hzで再サンプリングしてみます。
- 全データを10分ごとのセグメントに分割し、それぞれのセグメントをftspecによりフーリエ変換し、その結果からPNS,
SNSを計算します。
filter /sl:0 /xc:1 /is:0.001 /ip:3,5 < sample4 > temp.1
lasttime=$(gawk -f lasttime.awk temp.1)
starttime=0
while((starttime <= lasttime))
do
((endtime = starttime + 10))
filter /sl:4 /xc:2 /ts:5 /bg:$(starttime) /ed:$(endtime) < temp.1 > temp.2
ftspec /sl:4 /ts:5 /lt /cg < temp.2 > temp.3
autons /cg /ai < temp.3 > temp.4
gawk -f result.awk temp.4 >> result.dat
((starttime = nowtime + 10))
done
参考書
- ローゼンバーグ 『Korn Shellプログラミング』
アジソン ウェスレイ・トッパン \4800
- 川村 『bash -- Manual & Reference --』 秀和システム \2400
- 植村、富永 『awkでプログラミング』
オーム社 \2800
- Dougherty 『sed & awk プログラミング』
アスキー出版局 \3800

